Recurso gratuito
Publicada la base de datos más grande hasta la fecha para entender las proteínas del cuerpo humano
DeepMind, el sistema de inteligencia artificial desarrollado por Google, y el Laboratorio Europeo de Biología Molecular (EMBL) publican los datos sobre 20.000 proteínas expresadas en el genoma humano
"Este es uno de los conjuntos de datos más importantes desde el mapa del Genoma Humano", argumenta Ewan Birney
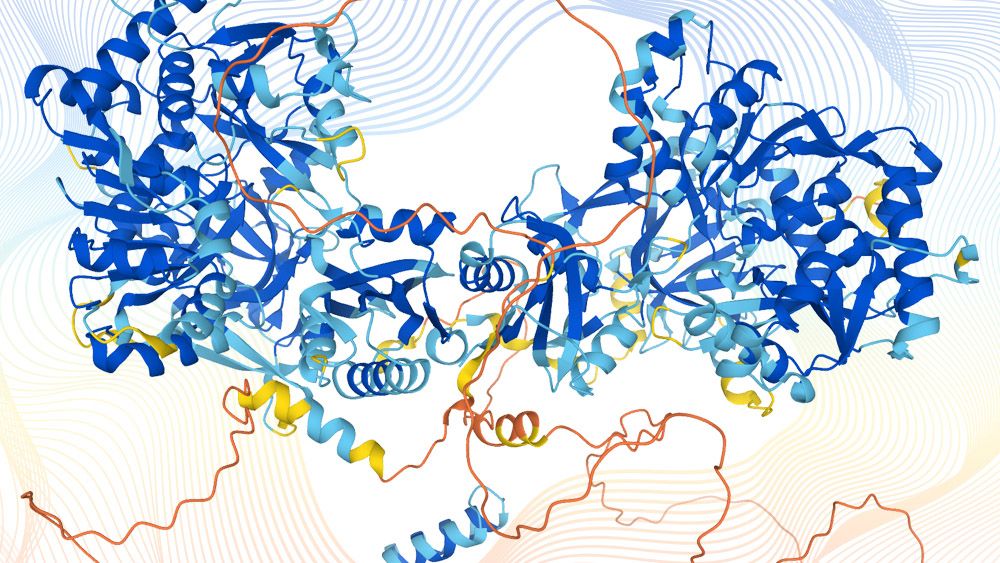
Representación artística de unas proteínas AlphaFold / Deepmind / AlphaFold / Deepmind


Valentina Raffio
Periodista.
Para entender cómo funciona el mundo, a veces, hay que empezar por lo más básico. Por las piezas sobre las que se construye todo lo demás. Muchos de los grandes descubrimientos científicos, de hecho, empiezan interpelando a las protagonistas ocultas sobre las que se construyen todas las formas de vida: las proteínas. Entender cómo funcionan, cómo interactúan con el entorno o cómo se pliegan resulta clave para tanto para descifrar enigmas de la biología y como para plantear soluciones efectivas ante problemas estructurales. Todo este complejo camino se simplifica, en parte, a partir de hoy con la publicación de la base de datos más completa hasta la fecha sobre predicciones de estructuras 3D de proteínas humanas.
DeepMind, el sistema de inteligencia artificial desarrollado por Google, y el Laboratorio Europeo de Biología Molecular (EMBL) anuncian este mismo miércoles la creación de esta iniciativa que proporcionará de manera gratuita y abierta los datos sobre 20.000 proteínas expresadas en el genoma humano. En la práctica, esta iniciativa se convertirá en una 'hemeroteca' para entender el 98,5% de todas las proteínas humanas. Este "tesoro de datos", a su vez, también proporcionará información muy valiosa para comprender los procesos biológicos, para entender la base de enfermedades raras y para guiar el desarrollo de tratamientos y fármacos.
La estrella de este anuncio es ni más ni menos que un algoritmo de inteligencia artificial de última generación: AlphaFold. La herramienta ha sido diseñada como un algoritmo de aprendizaje automático (o deep-learning) que, lejos de quedarse exclusivamente con las instrucciones de los programadores, aprende y mejora sobre la marcha. En diciembre del año pasado, DeepMind anunció que su algoritmo había logrado predecir de manera rápida y precisa la forma tridimensional de las proteínas humanas. Y esto, en palabras del biólogo evolutivo Andrei Lupas a la revista ‘Nature’, significaba "cambiarlo todo". Más ahora, que esta información será accesible para toda la comunidad científica.
"Este será uno de los conjuntos de datos más importantes desde el mapa del Genoma Humano", argumenta el director general de EMBL, Ewan Birney. "Hacer que las predicciones de AlphaFold sean accesibles a la comunidad científica internacional abre muchas nuevas vías de investigación, desde enfermedades desatendidas hasta nuevas enzimas para la biotecnología", valora Birney en un comunicado de prensa emitido por su centro.
Cincuenta años de enigmas
La comunidad científica lleva al menos cincuenta años inmersa en el 'problema del plegamiento de las proteínas'. De hecho, como decíamos hace un momento, entender la estructura de estas macromoléculas es clave para descifrar la causa estructural de enfermedades y todo tipo de dolencias. ¿El problema? Que esta pregunta, lejos de ser sencilla de responder, a menudo requiere meses o años de investigación. Y muchas veces ni así se consigue resolver. La irrupción de la inteligencia artificial en este proceso podría desatascar enigmas irresueltos y, a la vez, ayudaría a avanzar más rápidamente la investigación científica.
Representación artística de unas proteínas. / AlphaFold / Deepmind
"La base de datos de AlphaFold (AlphaFold Protein Structure Database) se basa en los descubrimientos de generaciones de científicos y científicas, desde los pioneros y las pioneras de la cristalografía y el análisis de estructura de las proteínas, hasta los miles de especialistas en predicción y biólogos y biólogas estructurales que han pasado años experimentando con proteínas desde entonces y que han compartido sus resultados de forma abierta", afirman los creadores de esta herramienta.
Hasta ahora, décadas de investigación solo habían logrado desentrañar el 17% de los aminoácidos del proteoma humano. El algoritmo de AlphaFold ha logrado avanzar hasta predecir el 58% de estas estructuras. En algunos casos, la inteligencia artificial proporcionará un pronóstico certero sobre estas proteínas para que, posteriormente, los científicos puedan certificar su estructura a través de experimentos. En otros casos, todo apunta a que la predicción no solo dará pistas sino que proporcionará "un grado de confianza muy alto". Esto, según sus creadores, ocurrirá en hasta el 35% de los casos.
El estreno de esta base de datos, anunciado a bombo y platillo por la revista 'Nature', además de presentar miles de estructuras de proteínas humanas, también ahondará en información sobre otros organismos clave en el avance científico. La ‘hemeroteca’ de AlphaFold, de hecho, albergará datos sobre 20 organismos utilizados como modelo de estudio como, por ejemplo, la bacteria E. coli, la mosca de la fruta, el ratón, el pez cebra, el parásito de la malaria y las bacterias de la tuberculosis. Ahora que toda esta información está a disposición del mundo, solo el tiempo dirá qué avances generará este recurso. Las posibilidades, al menos sobre el papel, son infinitas.
- Sumergir los pies en vinagre, la nueva tendencia que arrasa: estos son sus beneficios
- Rosalía disfruta de una cena familiar en un famoso restaurante del Born
- Illa replica a Aragonès y Puigdemont: "El uso del catalán ha retrocedido en una década de gobiernos de Junts y ERC
- Un final bochornoso en el Bernabéu
- El gran secreto para acabar con los pececillos de plata
- Olvídate de freír el huevo en la sartén: esta es la fórmula para hacer los mejores huevos fritos
- Álex ya está en casa: "Las Fuerzas Armadas nunca dejan a nadie atrás
- Este es el pueblo de Catalunya que participará en Grand Prix este verano
9 razones para viajar a Malta
